Recuento de células en ensayos de migración con ImageJ
Esta nota de aplicación describe un método para medir la migración celular, utilizando ImageJ, contando el número de células que han migrado a la Zona de Detección en un Ensayo de Migración Celular Oris. ImageJ es un programa gratuito de análisis de imágenes desarrollado en los Institutos Nacionales de Salud.
La migración celular es fundamental para muchos procesos fisiológicos, como el desarrollo embrionario, la cicatrización de heridas y la respuesta inflamatoria. Además, el comportamiento móvil aberrante de las células contribuye a procesos patológicos como la metástasis tumoral y la artritis (1).
En Ensayo de migración celular Oris (Figura 1) utiliza una placa de 96 pocillos con tapones de silicona que excluyen las células de la zona de detección central del pocillo. Después de sembrar las células y dejar que se adhieran, se retiran los tapones de silicona para dejar al descubierto una región no sembrada de 2 mm de diámetro en el centro de cada pocillo, a la que se permite migrar a las células.
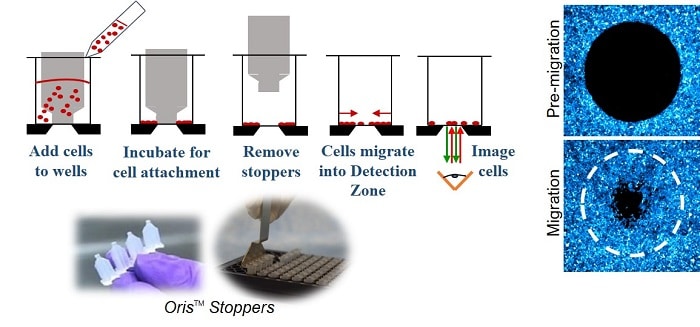
MÉTODOS
Las células epiteliales de mama MDA-MB-231 y las células de fibrosarcoma HT-1080 se cultivaron en una placa Oris™ Cell Migration Assay - TriCoated pocillos tratados con cultivo de tejidos, recubiertos con colágeno I o recubiertos con fibronectina. Después de 16 horas, las células se fijaron con glutaraldehído 0,25% y los núcleos celulares se se tiñeron con DAPI 1:2000 (Pierce). Las imágenes se adquirieron con un objetivo de 5X en un microscopio invertido Zeiss Axiovert 200 equipado con una cámara CCD.
La migración celular a la zona de detección se midió contando el número de células mediante el software de análisis ImageJ (versión 1.42l). En primer lugar, se fijó el para cada imagen en escala de grises (Imagen-> Ajustar-> Umbral). Al seleccionar "Aplicar" en la ventana la imagen umbralizada se convirtió en una imagen binaria. Los núcleos ligeramente superpuestos de Los núcleos ligeramente superpuestos se separaron mediante un proceso de segmentación Watershed (Proceso-> Binario-> Cuenca).
A partir de la imagen binaria, se creó una región de interés (ROI) circular de 2 mm de diámetro (el mismo diámetro que la punta del tapón). mediante el comando de menú Edición-> Selección-> Especificar. En la ventana Especificar, la "anchura" y la "altura" se fijaron en 2 mm, y se marcó la casilla "oval". se marcó. La ROI se centró sobre la zona de detección dentro de cada pocillo. En El número de núcleos contenidos en la ROI se cuantificó mediante el comando de menú Analizar-> Analizar partículas. Los valores para definir el tamaño mínimo y máximo eran 100 y 1000 píxeles2 , respectivamente. Se seleccionó "Mostrar máscaras" para mostrar un dibujo de los objetos detectados. Se seleccionaron "Resumen" y "Excluir en bordes" para el análisis.
Los recuentos celulares de la ventana de resumen (es decir, los recuentos) se exportaron a Windows Excel para su análisis estadístico. El número de núcleos para cada condición se promedió a partir de 8 pocillos.
RESULTADOS
En esta nota de aplicación, se evaluó la migración de células MDA-MB-231 y HT-1080 en tres superficies (tratada con cultivo de tejidos, colágeno I o fibronectina; Oris™ Cell Migation Assay - TriCoated), contando el número de células en la zona de detección mediante ImageJ. Las células MDA-MB-231 mostraron diferentes grados de migración a la zona de detección en función del recubrimiento de la superficie del pocillo (Figura 2). Las imágenes de fase de las células adquiridas inmediatamente después de la retirada del tapón (control de migración) y 16 horas después de la retirada del tapón demuestran diferencias en la migración celular dependiendo de si las células se sembraron en una superficie tratada con cultivo tisular, una superficie recubierta con colágeno I o una superficie recubierta con fibronectina (Figura 2A-D).
Utilizando ImageJ, se contaron las células marcadas con DAPI creando una región de interés (ROI) circular de 2 mm de tamaño similar a la zona de detección inicial (Figura 2E-H). Al realizar la función de análisis de partículas en ImageJ se obtuvieron dibujos de los objetos detectados que se contaron dentro de la ROI circular (Figura 2I-L). Las diferencias en la migración de células MDA-MB-231 a la zona de detección (ROI) se resaltaron superponiendo el dibujo del análisis de partículas a la imagen fluorescente original (Figura 2M-P).

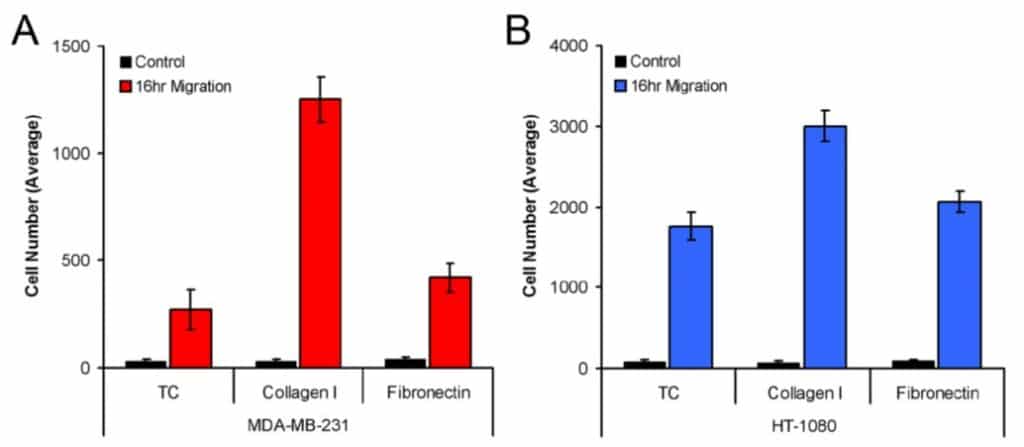
La figura 3 muestra el número medio de células MDA-MB-231 (3A) y HT-1080 (3B) que migraron a la zona de detección cuando se sembraron en pocillos tratados con cultivo de tejidos, recubiertos con colágeno I y recubiertos con fibronectina. Tanto las células MDA-MB-231 como las HT-1080 mostraron la migración más robusta en el colágeno I. Además, este método de análisis produjo diferencias estadísticas en la migración de las líneas celulares modelo en los tres recubrimientos de la placa (es decir, la migración de MDA-MB-231 en el colágeno I frente a la fibronectina).
CONCLUSIONES
Esta nota de aplicación demuestra un método para medir la migración celular en el Ensayo de Migración Celular Oris mediante el uso del software de análisis ImageJ para el recuento de células. Este estudio, que utiliza ImageJ para cuantificar el número de células en la zona de detección, demuestra que tanto las células MDA-MB-231 como las HT-1080 mostraron diferencias estadísticamente significativas en la migración cuando se sembraron en pocillos tratados con cultivo de tejidos, recubiertos con colágeno I y recubiertos con fibronectina. Utilizando el método de análisis detallado descrito aquí, ImageJ puede proporcionar una medida precisa de la migración celular cuando se utiliza el Ensayo de Migración Celular Oris.
Más información Ensayos de migración celular Oris.